ヘルスケア・ライフサイエンス分野において、大規模なオミクスデータを分析するためのコンピューティングのニーズは日々増大しています。今回は、AWS が提供するオミクスデータ分析のためのマネージドサービス「AWS HealthOmics」を使ったオミクスデータ分析ワークフローの構築方法について解説します。
AWS HealthOmics とは
AWS HealthOmics は、オミクス (ゲノム (Genomics) や転写産物 (Transcriptomics) など、生物学分野で -omics という名前を持つ大規模データの総称) データを扱うためのマネージドサービスです。オミクスデータを保存する Storage、クエリや分析を行う Analytics、分析ワークフローを実行する Workflows の 3 つの機能を提供しています。各機能の詳細については公式ページや AWS Blog の記事、公式ハンズオンなどをご参照ください。
AWS では、AWS HealthOmics による分析ワークフローの実行と分析結果の閲覧ができる Web アプリケーション「AWS HealthOmics Analysis App」をサンプルとして公開しています。今回はこのアプリケーションを題材に、AWS HealthOmics を使ったオミクスデータ分析ワークフローについて解説します。
AWS HealthOmics Analysis App の概要
AWS HealthOmics Analysis App は、AWS HealthOmics を使ってオミクスデータの分析ワークフローを実行し、分析結果を閲覧する Web アプリケーションのサンプル実装です。このアプリでは、以下のような機能を実装しています。
- AWS HealthOmics によるバイオインフォマティックスのワークフローの実行
- AWS Step Functions によるワークフローの二次解析 (可視化) や完了通知の実行
- Amazon Simple Email Service (SES) によるワークフロー完了通知メールの送信
- ワークフロー実行結果のブラウズ
- ワークフローで実行されたタスクの詳細情報の表示
アプリケーションの全体アーキテクチャと、各サービスの役割は以下の通りです。
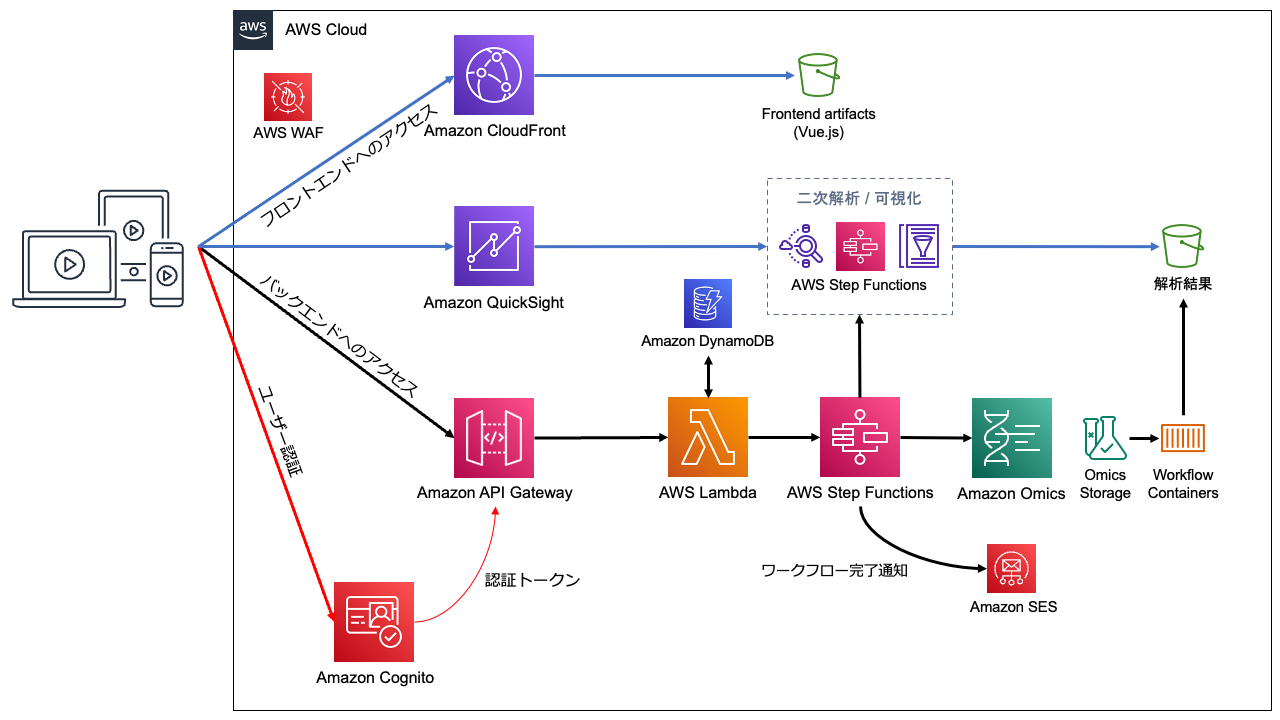
| サービス | 役割 |
|---|---|
| Amazon CloudFront | Vue.js で実装されたフロントエンドのアセットを配信 |
| Amazon S3 | Vue.js で実装されたフロントエンドのアセットを格納 |
| Amazon Cognito | ユーザー認証を行うためのユーザープールを提供 ユーザープールをフロントエンドやバックエンドから利用するためのユーザープールクライアントを提供 |
| Amazon API Gateway | フロントエンドに対してバックエンドの REST API を提供 |
| AWS Step Functions | AWS HealthOmics ワークフローの実行や二次解析 (可視化) 等のタスクを実行するステートマシンを提供 |
| AWS Lambda | バックエンド API を実装した Lambda 関数を実行 Step Functions ステートマシンのタスクを実装した Lambda 関数を実行 |
| Amazon DynamoDB | Step Functions で実行される二次解析 (可視化) の実行結果を管理 |
| Amazon QuickSight | ワークフローの出力を可視化するためのダッシュボードを表示 |
| AWS WAF | フロントエンドやバックエンド API にアクセスできる IP アドレスを制限 |
GitHub の README に書かれたデプロイ手順に従って、利用方法を解説します。
AWS HealthOmics Analysis App のデプロイ
前提条件
AWS HealthOmics Analysis App は AWS Cloud Development Kit (CDK) を使って AWS サービスの設定を行います。AWS CDK を使うには、前提条件として以下のソフトウェアがインストールされている必要があります。詳細はこちらのドキュメントをご参照ください。
上記の条件を満たす環境として、AWS Cloud9 を使う事もできます。
デプロイ
AWS HealthOmics Analysis App の GitHub リポジトリを clone し、デプロイ手順のドキュメントに従ってデプロイを実施してください。
なお、AWS HealthOmics が利用できるリージョンは現時点 (2023/9/20) で以下のようになっており、AWS HealthOmics Analysis App は us-east-1 リージョンにデプロイされる事を前提としています。
- us-east-1 (米国東部: バージニア北部)
- us-west-2 (米国西部: オレゴン)
- ap-southeast-1 (アジアパシフィック: シンガポール)
- eu-central-1 (欧州: フランクフルト)
- eu-west-1 (欧州: アイルランド)
- eu-west-2 (欧州: ロンドン)
Ready2Run ワークフローを実行する
デプロイが完了したら、早速ワークフローを実行してみましょう。AWS HealthOmics では、以下の2種類のワークフローを実行することができます。
- 初期設定不要ですぐに使える Ready2Run ワークフロー
- ユーザーがコードと初期設定を任意にカスタマイズできるプライベートワークフロー
今回は、Ready2Run ワークフローの中から、タンパク質の立体構造を予測する AlphaFold を実行しようと思います。Ready2Run ワークフローの使い方は AWS HealthOmics コンソールの Ready2Run ワークフローのページで確認する事ができ、簡単に実行を試せるサンプルデータも提供されているので、今回はそれを利用して AlphaFold を実行します。
実行手順
- AWS HealthOmics Analysis App にサインインしたら「New Analysis」をクリックします。
- 次の画面で「Workflow」をクリックすると Ready2Run ワークフローの一覧が表示されるので、「AlphaFold for up to 600 residues」を選択します。
- 「Analysis Name」に任意の名前を入力して「CONTINUE」をクリックします。(「Visualizer」は今回は指定しません)
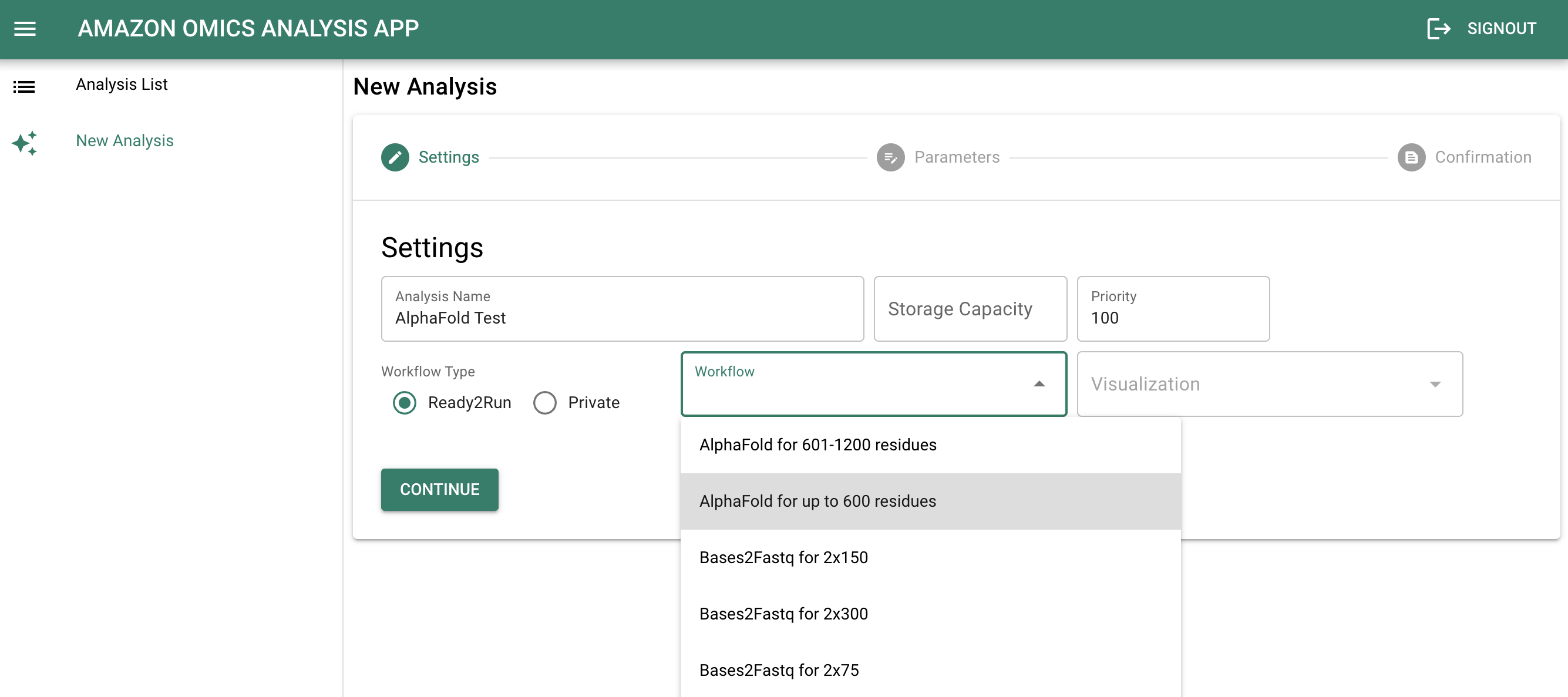
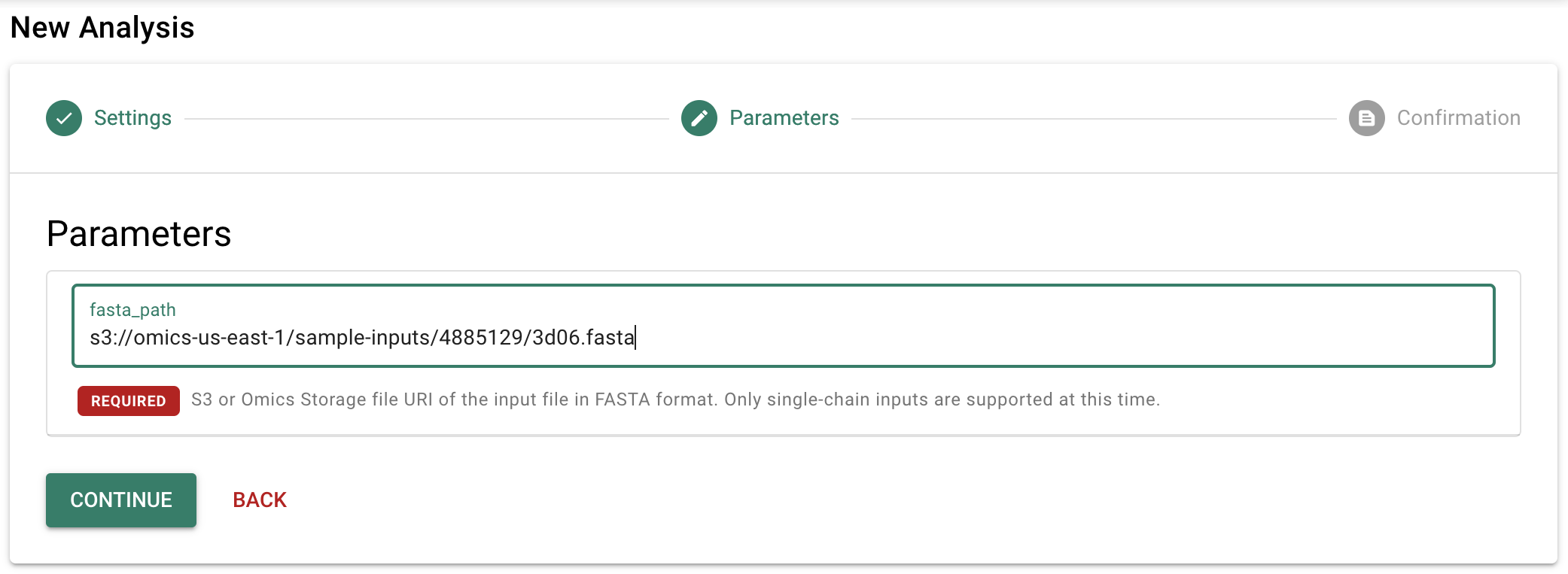
- 次の画面で「fasta_path」に以下の S3 URL を入力し、「CONTINUE」をクリックします。
s3://omics-us-east-1/sample-inputs/4885129/3d06.fasta
- 次の画面に表示された内容を確認し、「RUN」をクリックします。
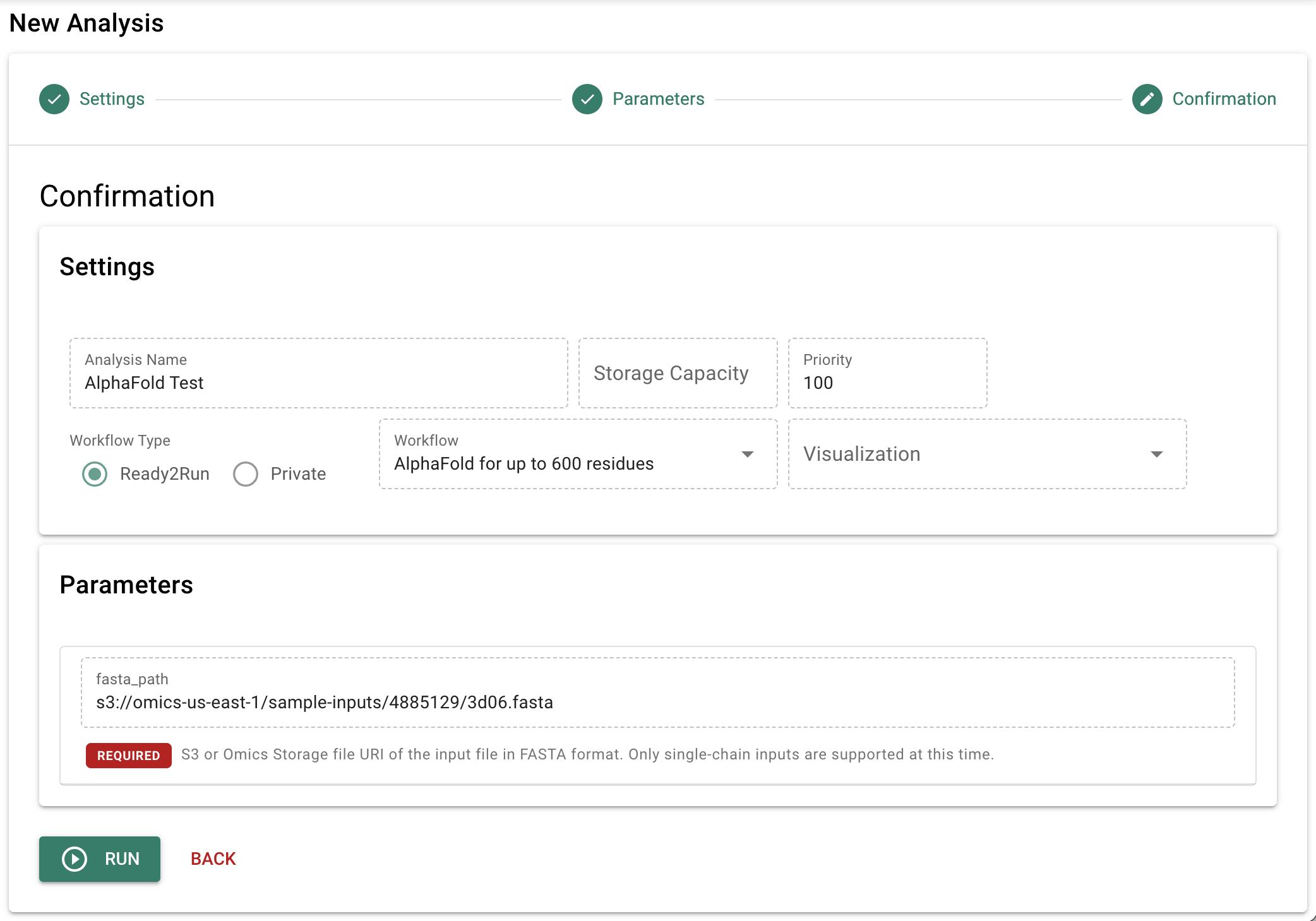
- 確認ダイアログが表示されるので、「OK」をクリックします。
入力内容に問題がなければ、ワークフローを実行するための Step Functions ステートマシンが開始されます。「Analysis List」の更新ボタンを押すと、開始された AWS HealthOmics ワークフローが一覧に表示されます。
AlphaFold のサンプルの実行には6〜7時間ほどかかりますが、完了するとアカウントのメールアドレスに完了通知メールが送信されます。
(メールを受信するには、事前にそのメールアドレスを Amazon SES の「検証済みID」として登録しておく必要があります。詳細は Amazon SES デベロッパーガイドの「Eメールアドレス ID の作成」をご参照ください。)
Step Functions によるワークフローの実行
Step Functions ステートマシンでは、以下のようなステップでタスクが実行されます。
- AWS HealthOmics ワークフローを実行する。
- (指定されていれば) 二次解析 (可視化) を実行する。
- 完了通知を実行する。
- ステートマシンの終了処理。
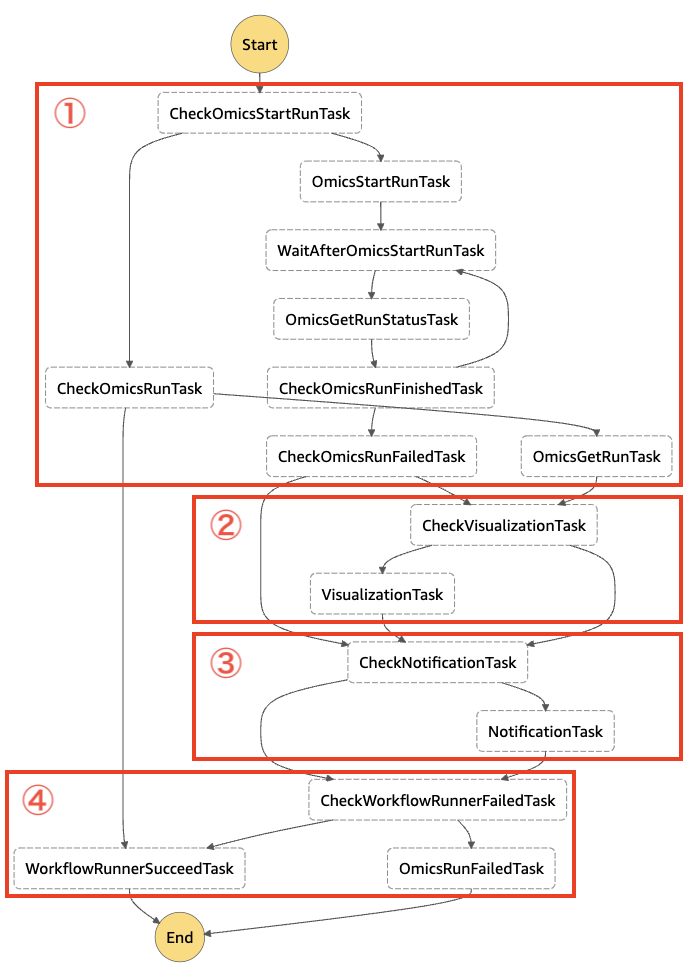
各ステップで実行されるタスクを解説します。タスクの詳細については公式ドキュメントをご参照ください。
AWS HealthOmics ワークフローの実行
| タスク名 | タスク種別 | 処理内容 |
|---|---|---|
| CheckOmicsStartRunTask | Choice | ステートマシンの入力に OmicsStartRun が含まれているかを確認 |
| OmicsStartRunTask | Lambda | AWS HealthOmics のワークフローを実行 |
| WaitAfterOmicsStartRunTask | Wait | AWS HealthOmics のワークフロー完了を数分間待機 |
| OmicsGetRunStatusTask | Lambda | AWS HealthOmics のワークフロー実行状態を取得し、OmicsRun として出力 |
| CheckOmicsRunFinishedTask | Choice | AWS HealthOmics ワークフローが完了したかを確認 |
| CheckOmicsRunFailedTask | Choice | AWS HealthOmics ワークフローが失敗したかを確認 |
ステートマシンの入力に OmicsStartRun が含まれていなかった場合、ワークフローの新規実行は行わずに以下のタスクに分岐します。
| タスク名 | タスク種別 | 処理内容 |
|---|---|---|
| CheckOmicsRunTask | Choice | ステートマシンの入力に OmicsRun が含まれているかを確認 |
| OmicsGetRunTask | Lambda | AWS HealthOmics のワークフロー実行状態を取得する |
二次解析 (可視化) の実行
| タスク名 | タスク種別 | 処理内容 |
|---|---|---|
| CheckVisualizationTask | Choice | ステートマシンの入力に Visualization が含まれているかを確認 |
| VisualizationTask | Step Functions | ワークフロー実行結果の二次解析 (可視化) を実行するため、別の Step Functions ステートマシンを起動 |
完了通知の実行
| タスク名 | タスク種別 | 処理内容 |
|---|---|---|
| CheckNotificationTask | Choice | ステートマシンの入力に Notification が含まれているかを確認 |
| NotificationTask | Lambda | Amazon SES で完了通知メールを送信 |
ステートマシンの終了処理
| タスク名 | タスク種別 | 処理内容 |
|---|---|---|
| CheckWorkflowRunnerFailedTask | Choice | AWS HealthOmics ワークフローが失敗したかを確認 |
| WorkflowRunnerSucceedTask | Succeed | ステートマシンの状態を正常終了とする |
| OmicsRunFailedTask | Fail | ステートマシンの状態を失敗とする |
分析結果のブラウズ
ワークフローの実行が完了したら、分析結果をブラウズする事ができるようになります。「Analysis List」で該当のワークフローをクリックするか、または完了通知メールが届いた場合はメール内のリンクをクリックすると分析の詳細情報が表示され、「Outputs」でワークフローの出力ファイルをブラウズすることができます。
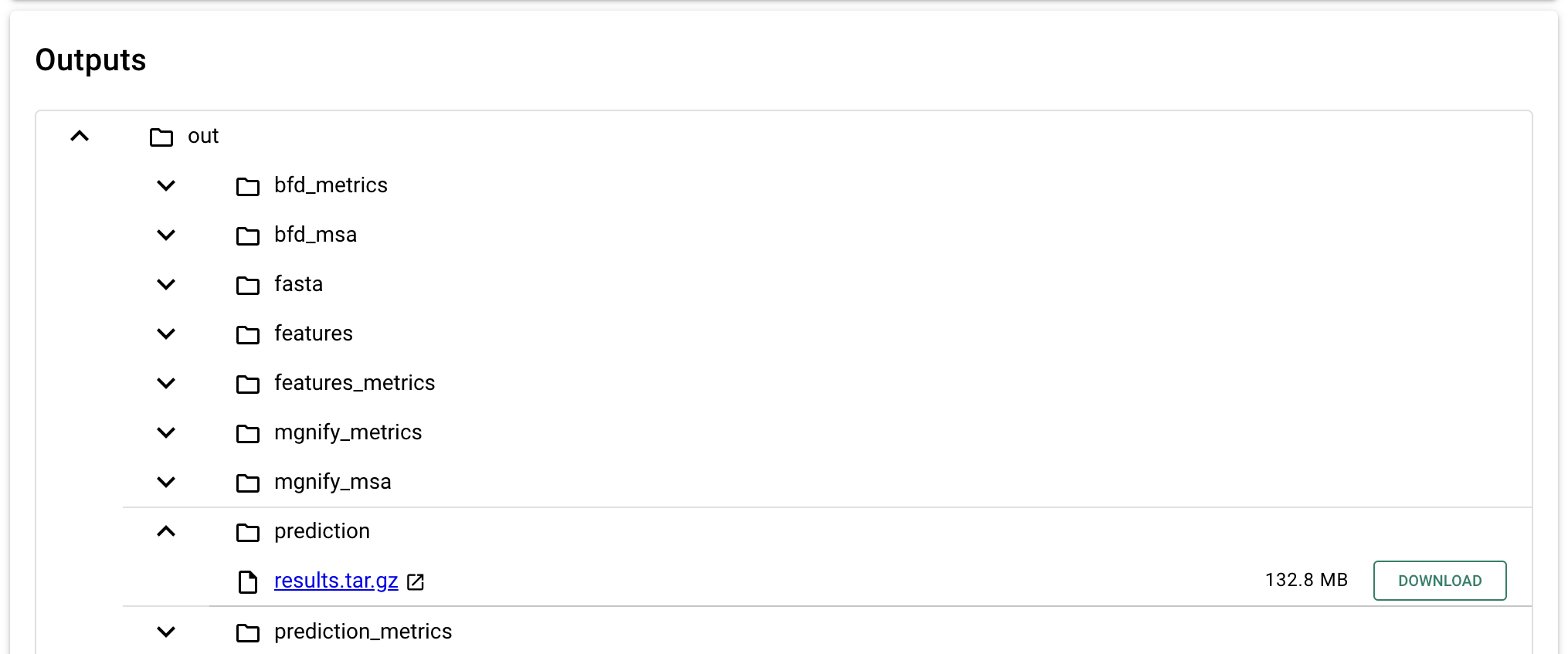
ファイル名をクリックすると、ブラウザで表示可能な形式 (PDF や画像など) のファイルであれば別のタブで表示されます。「DOWNLOAD」をクリックすると、ファイルをダウンロードすることができます。
ブラウザで表示できない形式の出力ファイルについては、AWS HealthOmics Analysis App に二次解析によるデータ変換や可視化手法の登録を行うことで表示が可能となります。詳細については別の記事で解説予定です。
「Tasks」では、ワークフローで実行されたタスクの詳細情報を確認することができます。
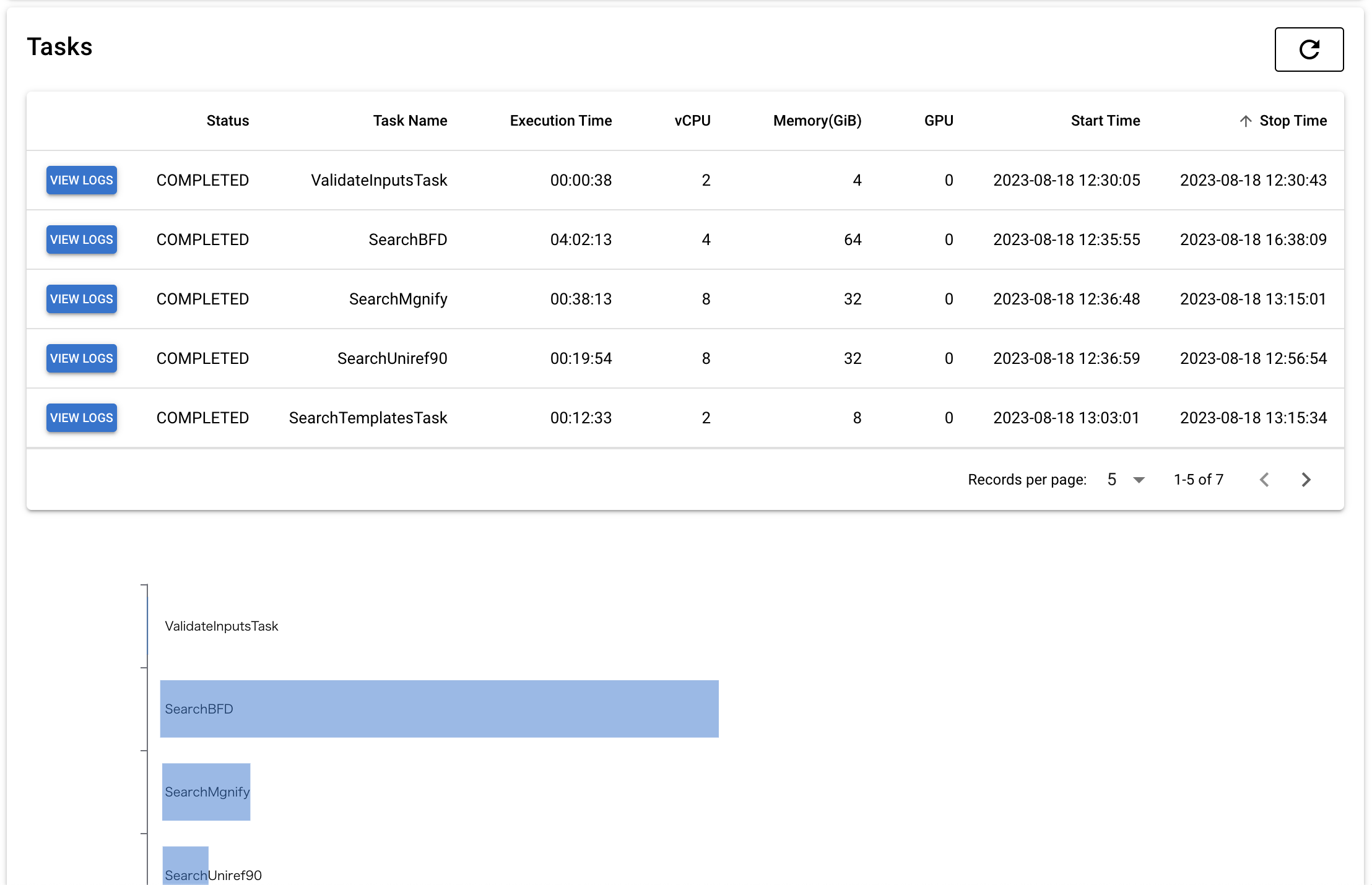
タスクの名前と実行時間、使用した vCPU やメモリ量などが表形式で表示され、その下では実行の時系列をグラフで確認できます。「VIEW LOGS」ボタンを押すと、タスクが実行中に CloudWatch Logs に出力したログを確認することができます。
まとめ
今回は、AWS HealthOmics のご紹介と、このサービスを AWS Step Functions と組み合わせたサンプルアプリである AWS HealthOmics Analysis App の解説を行いました。これらのサービスを活用することで、スケーラブルなオミクスデータ分析ワークフローを AWS 上に手軽に構築する事が可能となります。
